低水平的“记忆蛋白”与阿尔茨海默氏症认知能力的下降
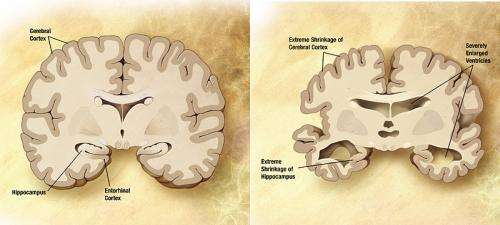
与人类大脑组织样本和基因工程小鼠,约翰霍普金斯大学的医学研究人员和美国国立卫生研究院的同事们一起,加州大学圣地亚哥Shiley-Marcos阿尔茨海默病研究中心,哥伦比亚大学和研究所基础研究在史泰登岛说后果的低水平的蛋白质NPTX2阿尔茨海默病(AD)患者的大脑可能会改变的神经活动模式的方式导致学习和记忆损失特征的疾病。
这一发现,在4月25日版的描述eLife,将导致重要的研究和有一天帮助专家开发新的和更好的治疗老年痴呆症和其他形式的认知能力下降。
目前广告影响了超过五百万的美国人。
团的蛋白质称为淀粉样斑块,看到广告的人的大脑中,往往与疾病相关的智力下降的罪魁祸首。但尸检和脑成像研究表明,人们可以有很高的水平的淀粉样蛋白没有显示广告的症状,质疑淀粉样蛋白和痴呆之间的直接联系。
这项新研究表明,当蛋白质NPTX2“拒绝”与此同时,淀粉样蛋白在大脑中积累,对神经元电路适应性是必不可少的“和谐说”中断,导致记忆的失败。
“这些发现是非常有趣的东西如何认知失败人类阿尔茨海默氏症,”Paul沃雷说,医学博士,约翰霍普金斯大学医学院的神经学家和文章的资深作者。“关键的一点是,它的淀粉样蛋白的结合和低NPTX2导致认知失败。”
自1990年代以来,沃利的小组一直在研究一组基因被称为所谓的“立即早期基因,”因为他们几乎立即激活脑细胞,当人们和其他动物有一个经验,结果在一个新的记忆。
NPTX2是其中的一个立即早期基因的基因被激活,使蛋白质神经元用来加强大脑的“电路”。
“这些联系是必不可少的对大脑建立同步组“电路”应对经验,”沃利说。“没有他们,神经元激活不能有效地同步和大脑不能处理信息。”
沃雷说他被先前的研究表明改变阿尔茨海默氏症患者的大脑活动模式。沃利的小组怀疑改变活动与立即早期基因功能的变化。
获得答案,研究人员首先将一个图书馆144年存档人脑组织样本来衡量NPTX2基因编码的蛋白质水平。NPTX2蛋白质水平,他们发现,减少高达90%的大脑样本广告相比同龄的人大脑样本没有广告。相比之下,与淀粉样斑块的人从来没有显示广告的NPTX2正常水平的迹象。这是一个初步的建议NPTX2和认知之间的联系。
之前的研究已经表明NPTX2起着关键作用发育大脑布线和抗实验性癫痫。研究李峰NPTX2水平可能与广告的认知功能障碍,沃利和他的同事研究了小鼠没有啮齿动物相当于NPTX2基因。
测试表明,缺乏NPTX2单独并不足以影响大脑细胞的功能的测试片。然后研究人员添加到老鼠的基因,增加大脑中的淀粉样蛋白生成。从小鼠大脑切片与淀粉样蛋白和没有NPTX2 fast-spiking中间神经元无法控制大脑“节奏”重要制造新的记忆。此外,谷氨酸受体通常是表示在中间神经元和基本功能是抑制中间神经元由于淀粉样蛋白和NPTX2删除鼠标和同样减少了人类大脑的广告。
沃雷说,结果表明,增加活动在AD患者的大脑是由于NPTX2低,加上淀粉样斑块,中间神经原的顺向中断函数。如果NPTX2和淀粉样蛋白的影响synergistic-one根据其他影响可以解释为什么不是所有人与大脑淀粉样蛋白表现出高水平的广告。
然后团队检查NPTX2蛋白质的脑脊液(CSF) 60生活AD患者和72人没有广告。低分数的记忆和认知标准广告测试,他们发现,与低水平的有关NPTX2 CSF。此外,NPTX2海马体的大小,与措施大脑内存区域的收缩在广告。在这种患者人群,NPTX2水平更紧密地与认知能力比目前最好biomarkers-includingτ,神经退行性疾病的生物标志物,被称为A-beta-42生物标志物,长期以来一直与广告有关。总的来说,NPTX2水平在AD患者的CSF 36比没有广告的人低70%。
“也许最重要的发现是,减少NPTX2似乎是独立的机制,生成淀粉样斑块。这意味着NPTX2代表了一个新的机制,强烈成立于基础科学研究,此前还没有研究在人类疾病动物模型的上下文中或。这就产生了许多新的机会,”沃利说。
“立即应用程序可能确定NPTX2的措施可以帮助病人的排序和识别的一个子集最适应新兴疗法。”Worley says. For instance, drugs that disrupt amyloid may be more effective in patients with relatively high NPTX2. His group is now providing reagents to companies to assess development of a commercial test that measures NPTX2 levels.
沃雷补充说,还需要更多的工作去理解为什么NPTX2成为广告的低水平和如何预防或减缓这一过程。
更多信息:肖Mei-Fang et al, NPTX2和认知功能障碍在阿尔茨海默氏症,eLife(2017)。DOI: 10.7554 / eLife.23798