研究小组发现了自闭症大脑出错的关键新线索

一个由加州大学洛杉矶分校领导的科学家团队发现了自闭症患者大脑问题的重要线索,自闭症是一种无法治愈的发育障碍,科学家对其成因也没有深入了解。
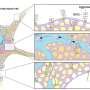
新的见解涉及到RNA编辑——在这种编辑中,遗传物质是正常的,但RNA的修饰改变了核苷酸,其模式携带了构建蛋白质所需的数据。
“RNA编辑可能在大脑中有重大的生理作用,但我们知之甚少,”合著者Daniel Geschwind博士说,他是加州大学洛杉矶分校的Gordon和Virginia MacDonald人类遗传学、神经病学和精神病学杰出教授,也是加州大学洛杉矶分校精确健康研究所所长。“RNA编辑是一个神秘的领域,其生物学意义还没有得到太多的探索。我们知道只有少数RNA编辑位点对蛋白质有什么作用。这项研究提供了一个新的关键线索来理解大脑中出错的地方自闭症病人。”
据估计,全世界有超过2400万人患有自闭症。截至2017年,在发达国家,约1.5%的儿童被诊断患有自闭症谱系障碍。这种障碍会影响交流和行为,其特征是在社会交流和社会互动方面存在问题,以及重复行为。
Geschwind说:“我们需要了解一系列遗传和环境因素是如何汇聚在一起导致自闭症的。”“RNA编辑是自闭症拼图的一个重要部分,但它完全被低估了。”
研究人员分析了69名死者的大脑样本,其中约一半人患有自闭症谱系障碍(包括自闭症和相关疾病),约一半人没有并担任过自闭症患者对照组.
该研究的资深作者、加州大学洛杉矶分校综合生物学和生理学的Maria R. Ross教授肖欣舒(Grace Xiao)和她的研究团队分析了每个大脑样本的70亿个核苷酸。
肖的团队发现自闭症患者的基因编辑能力下降。具体来说,他们在自闭症患者的大脑额叶皮层中确定了3314个编辑位点,这些位点的RNA编辑水平与对照组不同。在肖的实验室工作的加州大学洛杉矶分校生物信息学跨部门项目的研究生Stephen Tran是第一作者,他说,在2308个位点中,自闭症患者的RNA编辑减少了。他补充说,在另外1006个基因中,他们的RNA编辑水平有所提高。
Tran说,在大脑的颞皮层,自闭症患者在2412个编辑位点上的RNA编辑水平与对照组不同,其中1471个位点的编辑水平下降。在大脑小脑中,自闭症组成员的4340个位点的RNA编辑水平与对照组成员不同,其中3330个位点的RNA编辑水平下降。这三个大脑区域在自闭症中都很重要。
这项研究发表在该杂志上自然神经科学,是第一个全面研究RNA编辑在自闭症谱系障碍。
肖说,RNA编辑可以被认为是RNA突变,类似于与许多疾病有关的DNA突变。
“同一段DNA可以产生多个版本的RNA,并可能导致不同的蛋白质序列,”加州大学洛杉矶分校生物信息学跨部门研究生项目主任肖说。“RNA编辑允许细胞创造DNA中没有的新蛋白质序列。”
长期以来,科学家们一直认为RNA序列是基因DNA序列的忠实副本,而RNA只是将DNA指令传递到细胞其他部分的细胞信使。“当RNA编辑在20世纪80年代首次被发现时,这种假设被证明是错误的,”肖说,“我们发现了许多例子,我们从父母那里继承的遗传密码在我们的细胞中被编辑。”
在另一项重大发现中,研究人员发现了两种蛋白质,称为FMRP和FXR1P,它们在自闭症谱系障碍中调节异常的RNA编辑。Tran发现,FMRP增加RNA编辑,而FXR1P减少RNA编辑。自闭症组受FMRP调控的编辑水平降低,总体上RNA编辑也减少。
肖说:“这是第一个强有力的数据,显示了FMRP和FXR1P在人脑和自闭症中广泛而直接的功能作用。”
“FMRP所做的某些事情显然对自闭症的发病机制至关重要,”Geschwind说。“Grace和她的团队表明,这两种相关的蛋白质可能是RNA编辑减少的原因,以及偶尔增加的RNA编辑。”
他说,目前还不清楚自闭症患者在RNA编辑方面的变化是导致他们自闭症的原因,还是导致了自闭症,或者是自闭症的结果。“我们不能确定因果关系,”Geschwind说,他称赞肖团队的研究“优雅而出色”。
在精神分裂症、躁郁症和重度抑郁症中,RNA编辑也可能被破坏。研究小组计划继续研究这种疾病以及其他脑部疾病。
肖和Tran通过分析另一组22人的额叶皮层,重复了他们的发现自闭症谱系障碍对照组有23人没有这种障碍。Tran说,他们发现了与最初发现的相同的编辑减少模式。
研究人员在与自闭症相关的关键神经学基因中发现了RNA编辑改变,包括CNTNAP2和CNTNAP4, NRXN1和NRXN3, ANK2, NOVA1和RBFOX1。
肖和Tran使用了强大的生物信息学和统计学方法来识别RNA编辑位点,其中包括肖和她实验室前博士后学者张庆在2015年设计的类似GIREMI的方法。
在寻找疾病的原因时,大多数研究都集中在寻找DNA中的突变。肖说:“直到最近,我们还没有找到DNA中没有编码的RNA突变。RNA的这些变化可能会产生与DNA突变类似的影响。”
研究人员说,这项研究可能最终会带来治疗自闭症的新疗法,但可能不会持续很多年。
进一步探索