窥视脑瘤的基因组
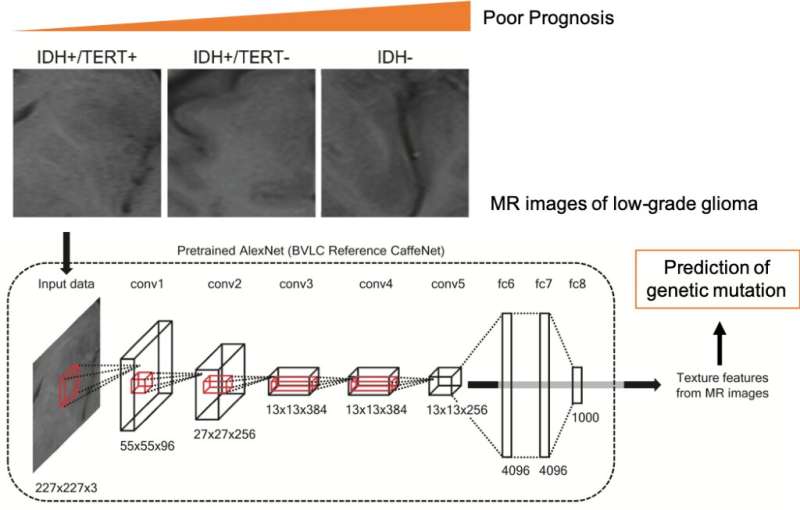
大阪大学(Osaka University)的研究人员开发了一种计算机方法,利用磁共振成像(MRI)和机器学习来快速预测发生在大脑或脊柱的神经胶质瘤肿瘤中的基因突变。这项工作可以帮助胶质瘤患者更快地接受更合适的治疗,从而获得更好的结果。这项研究最近发表在科学报告。
近年来,癌症治疗经历了一场革命。我意识到每个癌症病例都是独一无二的基因突变肿瘤细胞携带的基因现在被测序,以发现哪种化疗药物效果最好。然而,某些类型的癌症,特别是脑瘤,很难进行基因检测。只有在手术过程中采集样本,才能发现肿瘤的基因型,这可能会严重延迟治疗。
胶质瘤是一种源于大脑支持细胞的癌症。两种类型的突变尤为重要;这些是酶异柠檬酸脱氢酶(IDH)或端粒酶(TERT)的启动子区的基因的变化。鉴定这些突变可以帮助指导适当的治疗过程。研究人员制作了一个机器学习算法仅仅通过肿瘤的MR图像就可以预测哪些突变出现。
“机器学习越来越多地被用于诊断医学图像。但我们的工作是第一批试图将基因型这样隐藏的东西分类的人图像数据研究第一作者福隈亮平解释道。研究发现,与常规使用的磁共振图像的放射学特征(如大小、形状和强度)相比,该算法在预测突变方面明显更好。
为了构建该算法,研究人员使用卷积神经网络从MR图像中提取特征。然后,使用一个机器学习将患者分类为基于突变的存在或不存在的患者将患者分为群体。“我们希望扩大这种方法的其他类型的癌症,因此我们可以利用已经收集的大型癌症基因数据库”高级作者Haruhiko Kishima说。
最终的结果可以消除手术组织取样的需要。更重要的是,随着提供个性化药物的过程变得更容易、更快,它可能会为患者带来更好的临床结果。
文章,“预测低级胶质瘤中的IDH和TERT启动子突变磁共振使用卷积神经网络的图像“发表在科学报告。
进一步探索











用户评论