研究人员使用可解释的深度学习预测来识别新药
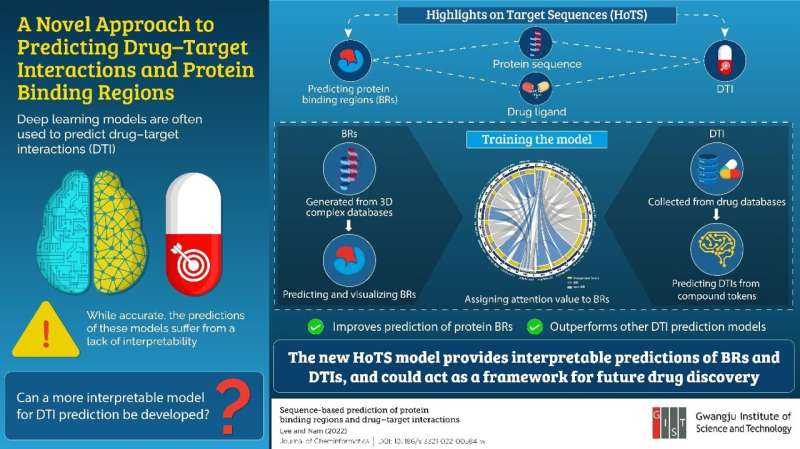
药物发现的一个重要方面是确定药物与其目标(蛋白质)分子的结合程度。通常,这一步包括将药物及其靶蛋白的3D结构以不同的配置进行对齐,这一过程称为“对接”。然后通过对特定目标分子的多种候选药物反复运行对接模拟来发现首选的结合位点。通常,由于潜在药物-靶点相互作用的可能候选者数量众多,因此使用深度学习模型来进行此类模拟。然而,使用这些模型的一个问题是很难解释它们的预测。虽然深度学习确实有助于快速发现药物,但它只不过是一个黑盒子。此外,对接模拟不能用于开发针对新靶点的药物,这些靶点没有已知的与相互作用药物的3D复合物。
最近的一项研究发表在化学信息学杂志来自韩国光州科学技术学院的副教授Hojung Nam和她的博士生Ingoo Lee开发了一种新的深度学习模型,称为“靶序列亮点”(HoTS),用于预测药物和靶分子之间的结合。这个新模型不仅能做出更好的预测,更有趣的是,它还能以一种可解释的方式进行预测。此外,该模型可以预测药物-靶点相互作用(DTIs),而不需要模拟或3D结构。
该团队是如何实现这一壮举的?南教授解释了他们的药物-靶点相互作用预测模型是如何工作的:“首先,我们使用先验知识明确地告诉模型蛋白质序列的哪些部分将与药物相互作用。然后利用训练好的模型识别和预测药物与靶蛋白之间的相互作用,具有更好的预测性能。利用这一点,我们建立了一个模型,可以预测靶蛋白的结合区域及其与药物的相互作用,而无需3d复合物。”
该模型不是处理蛋白质序列的完整长度,而是仅根据蛋白质中与DTI相互作用相关的部分进行预测。南教授解释说:“我们教模型‘关注’哪里,以确保它能够理解蛋白质的重要子区域,从而预测其与候选药物的相互作用。”反过来,这使得该模型能够比现有模型更准确地预测DTIs。
考虑到该模型不需要关于3D结构的信息,并且提供了清晰易懂的预测,本研究的发现为进一步的对接模拟预测新的模型提供了一个很好的起点候选药物针对靶向治疗,加速这一领域的发展。“我们研究中使用的模型将使药物发现过程更加透明,风险更低,成本更低。这将使研究人员在相同的预算和时间内发现更多的药物。”南教授总结道。
更多信息:Ingoo Lee等人,基于序列的蛋白质结合区域和药物-靶点相互作用预测,化学信息学杂志(2022)。DOI: 10.1186 / s13321 - 022 - 00584 - w