深度学习能够发现癌症的新基因突变
发表在《柳叶刀》杂志上的一项研究称,一种机器学习模型帮助科学家发现了目前基因组测序无法检测到的数百种癌症基因突变科学的进步.
这些发现提供了新的目标癌症冯岳博士是Duane和Susan Burnham分子医学教授,也是该研究的资深作者。
“我们的工作确定了许多以前未知的核聚变事件癌症基因组并且还捕获了已知致癌基因的新调控机制,”Yue说,他也是西北大学罗伯特·h·Lurie综合癌症中心生物化学和分子遗传学副教授,病理学和癌症基因组学中心主任。
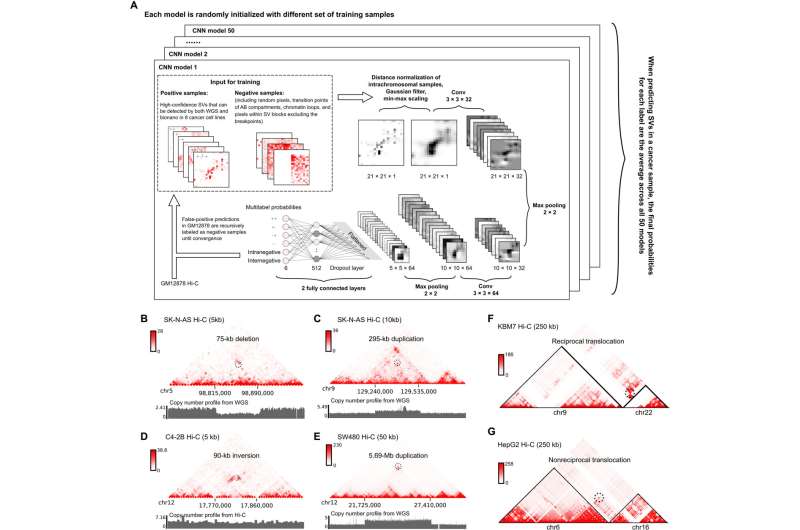
在每个细胞内,长链的DNA需要被精确地折叠和组织起来,这样它们才能适合直径通常只有几微米的细胞核。之前,岳和他的合作者已经证明了这一点结构变异在癌症基因组中,如倒置或易位,可以在基因组分析工具如Hi-C中检测到。
这些模式可以被计算机算法识别为结构变化的指标。此外,据Yue说,这些巨大的结构变异通常被全基因组测序(WGS)甚至长读测序(如Nanopore)遗漏。
“WGS非常擅长检测碱基对突变和短插入或缺失,但很难检测更大的变异,”Yue说,他也是医学增强智能研究所高级分子分析中心的主任。

在这项研究中,Yue和他的合作者从八种癌细胞系中收集了一组不同类型的高置信结构变异。这些是用来训练深度学习模型——命名为eaglec——学习隐藏在这些信号中的隐藏模式。结果与传统的基因组测序技术基本一致,通过WGS或Nanopore测序也发现了70% - 80%的基因组变异。
然而,EagleC发现了数百个被全基因组测序或长读测序遗漏的融合事件。岳说,这些新发现的事件占Hi-C检测到的全部遗传变异的10%到20%。
许多融合事件导致癌基因和远端增强子之间的连接,通常位于另一条染色体上。这些事件被称为“增强子劫持”,可导致致癌基因上调。
在这项研究中,研究人员使用EagleC搜索了100多个基因的结构变异癌细胞系以及患者样本,发现全基因组测序可能遗漏的其他融合事件。根据Yue的说法,使用这个模型可以扩展结构变异及其对癌症相关基因影响的知识。这对前列腺和乳腺癌这两种最常见的癌症也有高频率的融合事件。
Yue说:“我们可以观察有融合事件的癌症和没有融合事件的癌症在治疗反应上是否存在差异。”“我们的发现还为癌症研究人员提供了许多控制基本致癌基因和途径的新型调控因子。”
EagleC也可以用于检测单细胞Hi-C分析中的结构变化,其中数据稀疏。这使得科学家能够检查单个癌细胞之间的异质性。
Yue表示,未来他希望将这一模型应用于更多的癌症样本,并寻找针对新癌症的潜在药物融合在当前研究中发现的事件。
进一步探索