新的计算机模拟破解了癌症耐药性之谜
伊马替尼,更广为人知的名字是格列卫(Gleevec),在21世纪初进入市场时被誉为一种“奇迹”抗癌药。尽管它在治疗早期慢性髓系白血病(CML)(一种在骨髓细胞中形成的罕见癌症)方面非常成功,但许多晚期患者都经历了由体内重要蛋白质突变引起的耐药性。
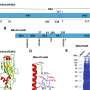
马里兰大学化学与生物化学系和物理科学与技术研究所的助理教授Pratyush Tiwary领导的一项新研究使用计算化学在分子水平上找出了导致格列卫耐药的原因。
这项研究发表在该杂志的网络版上安吉万德化学国际版2022年4月29日。杂志编辑给它贴上了“热门论文”的标签,这是一种授予最有兴趣和最重要的论文的荣誉。Tiwary的实验室开发的度量标准可能会有广泛的应用制药行业这可能会产生针对多种疾病的药物,而且疗效更高。
Tiwary说:“这项工作真正做的是让我们弄清楚哪些突变会使任何给定的药物无效,以及为什么。”“这些通常不能仅仅通过观察结构来预测蛋白质.你需要我的团队做的这些复杂的模拟来研究这个问题。在物理化学这就是所谓的热力学与动力学之争。”
在这项研究中,Tiwary和他的合著者专注于格列卫,它通过阻断一种被称为激酶的特定蛋白质来控制癌症。在某些情况下,激酶可能因药物阻断而发生突变,也可能因环境因素而发生突变,或者仅仅是偶然发生的。
Tiwary解释说:“在某种意义上,它类似于COVID,突变发生在刺突蛋白中。”“蛋白质的一小部分会变成其他东西,使药物无效。”
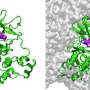
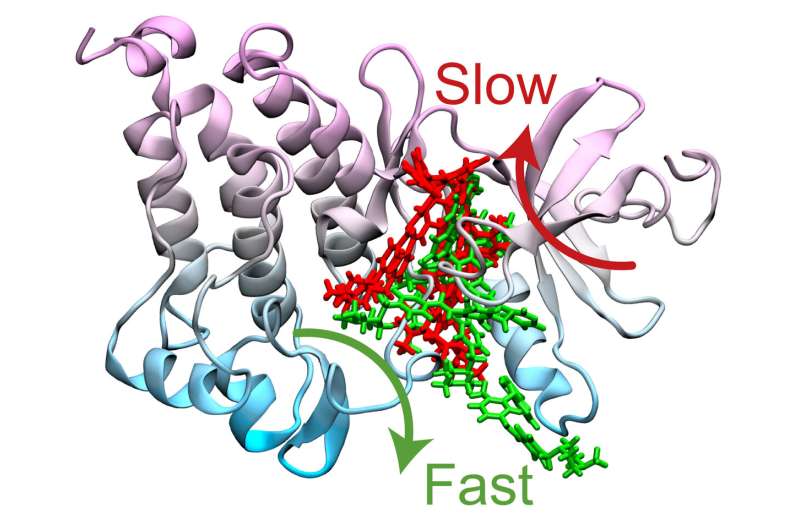
格列卫与一种蛋白质结合的地方发生的突变(称为药物结合袋)可以更清楚地解释其中的原因耐药性发生。然而,Tiwary和他的合著者开始回答一个更棘手的问题:为什么一种叫做N368S的特殊突变会引起耐药性,即使它似乎没有影响药物结合的方式?
这就是超级计算机的用武之地。Tiwary的实验室使用过机器学习化学的一个分支学科叫做统计力学设计计算机模拟方法,能够在几分钟、几小时甚至几天的时间内以极高的分辨率检查极小的结构——比如原子和分子。这是独一无二的,因为在大多数情况下,超级计算机只能以如此高的分辨率捕获微秒级的数据。
该模型通过轮流进行机器学习来估计格列卫可能离开蛋白质的途径。这些信息用于执行新的模拟,每一轮模拟都产生新的数据,训练机器学习模型学习改进的路径。这个过程会一直持续下去,直到机器学习和模拟彼此一致为止。
模拟最终确定了描述格列卫如何从蛋白质及其N368S突变体中解结合的两种最佳途径。Tiwary和他的合著者发现,N368S突变使蛋白质变得非常灵活,以至于其中一条通路打开,使格列卫在蛋白质开始工作之前就离开了蛋白质。
Tiwary的方法也可以反过来使用计算机模拟预测一种药物的变化,使其更有效,结合时间更长,这是他实验室的长期目标。另一项最近的研究发表在ACS中央科学, Tiwary的模拟被用来预测突变可能会影响RNA和小分子之间的相互作用。虽然计算机模拟可能永远不会取代传统的药物试验,这项新研究被称为“热门论文”表明计算化学在研究界赢得了应有的地位。
“无论是好是坏,在化学领域有一种心态,即模拟可能不是真正的化学。有时这是根深蒂固的。”Tiwary说。“这项工作表明,模拟可以补充实验,最终帮助我们解决与癌症等全球社会相关的巨大问题。”
进一步探索
王一航等,rna -小分子相互作用的结构探测与人工智能增强分子模拟,ACS中央科学(2022)。DOI: 10.1021 / acscentsci.2c00149