使用人工智能预测COVID激增

一组研究人员最近开发了一种人工智能模型,可以预测哪些冠状病毒变体可能会占主导地位并导致疫情激增。这项工作由哈佛医学院和马萨诸塞州总医院的医学助理教授Jacob Lemieux和麻省理工学院和哈佛大学Broad研究所的成员、哈佛大学文理学院的有机体和进化生物学教授、哈佛大学陈曾氏公共卫生学院的免疫学和传染病学教授Pardis Sabeti领导。它还受益于人工智能研究人员Fritz Obermeyer和Martin Jankowiak的工作,他们于2020年从优步人工智能实验室加入Broad,在那里他们开发了一种可以处理大量数据的机器学习模型,并为最新的工作提供了基础。
《公报》采访了Lemieux和Sabeti关于新的AI/机器学习模型它被称为PyR0(派- r -零),以及它将如何帮助应对当前的大流行和未来的疾病。
问答:Jacob Lemieux和Pardis Sabeti
宪报:你和同事们开发了一个机器学习模型,预测了至少两种传染性特别强的SARS-CoV-2变体的出现,这些变体在全球范围内引起了很多疾病。你能给我们介绍一下吗?
勒米厄:这个模型做出的最清晰的预测是,在欧米克隆子谱系中,BA.2是最适合的。在我们分析数据的时候,这是一种BA.1(欧米克隆)流行病,BA.1是每个人都关注的变异——最初是在南非,然后是世界上其他地方。该模型对BA.2更适合做出了相当强而自信的预测。这是基于BA.2在几个地方的动态,主要是印度和丹麦,事实证明这是相当准确的。从那时起,BA.2几乎在所有地方都取代了BA.1, BA.4或ba . 5实际上是BA.2的分支。这是对该模型至少预测动态的能力投下的信任票。
我们还进行了一项分析,回顾该模型在不同地区和全球范围内将会发生的情况。这个模型会发现α变种,B.117,也会发现δ,大约在同一时间,这些谱系是由领导的,高度协作的,非常劳动密集型的监视工作发现的。所以,我们认为这是一种补充,但不能取代人们对数据的关注,并在个别地区拟合重点模型。它的好处是它可以一次计算所有的数据,并跨地区汇总信息,这是一个人很难做到的。在这方面它是一个有用的工具。
宪报:随着BA.4和BA.5在今年夏天的发布,最近的运行告诉你关于即将到来的大流行的过程是什么?
勒米厄:该模型目前表明,BA.2.75值得关注,尽管它不认为与其他循环变体相比,BA.2.75的适应度差异太大。这表明ba 2.75可能会在某些地方接管,但可能不会在很大程度上改变疫情。
宪报:有没有提到疾病的严重程度?
LEMIEUX:没有。生长速度只是微生物的一种表型。但是还有很多其他的微生物表型,比如疾病的严重程度,可能也有遗传基础,希望我们能够用这样的方法弄清楚。在这个领域已经有很多关于耐药性的工作,我们已经在微生物基因型和微生物表型之间找到了很好的联系。所以,我乐观地认为,随着数据规模的增长,新的算法工具和计算能力的增强,我们将能够解决其中的一些问题。
宪报:我认为你分析的600万个基因组的数字会让大多数读者感到惊讶,如果你说的是独特的基因组的话。有多少个?
勒米厄:我们所说的基因组通常是来自个体患者的序列。我们倾向于认为一个基因组代表一个病人的病毒。这是数据库中数据的一个很好的近似。但每个患者的感染都对应着数百万个病毒副本,所以这只是大流行中发生的SARS-CoV-2复制事件数量的一小部分。
宪报:每个病人体内的病毒是否都有微小的变异?
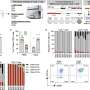
勒米厄:在一个特定的人身上有一些小的变化,但我们不需要把它们都建模来了解大流行。事实上,在共识水平上,不同个体之间的许多病毒序列是相同的。所以不存在650万个独特的基因组序列。有些是完全相同的。这实际上就是我们所追踪的,我们甚至将数据粗化[概括]到谱系的水平,这基本上是我们一起考虑的基因相似的基因组组。然后我们问,随着时间的推移,在不同的人群中:随着时间的推移,我们看到的那组被称为“谱系”的基因组是多了还是少了?为了这个模型的目的,我们使用了3000个谱系,每个谱系都包含一个独特的突变群。然而,这些突变可能发生在不止一个谱系中。这就是我们能够获得权力去询问哪些突变负责一个谱系随着时间的推移而增长或消亡。而且,由于世界各地的人们都在向这些数据库提供基因组,我们基本上可以实时了解哪些谱系在哪些地方生长,有时是由于随机的机会,就像一个大型的超级传播事件。 But if we find that the same lineage is dominating in Massachusetts and New York and California, that tells us there's probably something about that lineage. We're able to infer what that is by doing the same thing for mutations. If we see a mutation like N501Y, for example, that is consistently found in lineages that tend to grow, then we think that there's something about that mutation that causes that lineage to grow in a population.
宪报:这个模型能预测未来可能出现的变异吗,或者它真的能与现有的基因组一起工作,为可能传播的数千个谱系进行分类吗?它真的能向前看说,“嗯,这里可能会发生变异。这会是个问题吗?”
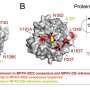
勒米厄:两者都有。它做得很好的一件事是提供了当前流通的不同谱系的增长率的估计。我们给在种群中观察到的每个突变分配一个适合度,如果一个突变以前从未被观察到过,我们就不能给它分配适合度。所以,如果有一种假设的菌株是由在其他地方观察到的突变组合而来的,但不是在同一个地方聚集在一起的血统以前,我们可以预测该菌株的增长率。如果我们没有观察到突变,模型就不知道那个特定突变的影响。
宪报:这项工作是如何开始的?
SABETI:雅各布,当时是一名医学院学生转博士后,和另一名研究生转博士后的丹尼·帕克,长期以来一直在研究检测微生物适应性变异的方法,从疟疾开始——这是实验室的一个激情项目。我们早期的工作是检测人类和其他哺乳动物的自然选择,挑战在于,因为世代时间太长,我们必须推断历史事件。在传染病,令人惊奇的是我们可以看到自然选择展现在我们眼前。我们可以实时追踪。这就是这种方法的力量。
但是当Jacob和其他人在十年前开始这项关于疟疾的研究时,数据实在是太少了。在埃博拉期间,我们开始获得高密度的数据,并与杰里米·鲁班(麻省大学陈医学院)一起发表了研究成果,确定了流行率上升的变异。但当时的数据仍然太少,无法对我们现在所能做的性质进行统计推断。随着大流行的到来,我们很快从没有足够的数据转变为我们有太多的数据以至于人们无法管理它。这是非常异构的数据:我们不知道数据来源;我们不知道这些序列的质量,也不知道如何管理和驯服这些庞大的数据集,以获得可靠的结果。
勒米厄:当时,我们还不习惯处理数以百万计的微生物基因组。我们习惯了处理成百上千的数据。那时我们开始与远大的PyR0团队合作,他们来自优步人工智能,在那里他们建立了这种概率编程语言来对非常大的数据集进行计算。Fritz Obermeyer是这个项目的主要负责人。他能够建立一个模型来解释哪些谱系在人群中传播得更容易,增长得更快,并用组成突变来表示这些谱系。Fritz工作的另一个重要创新是,它可以在现代处理硬件上运行,利用软件工程和现代计算能力的创新。这使得这一切以一种以前不可能的方式成为可能。
宪报:跨学科方法在这项研究中有多重要?听起来你牵扯了很多不同的人。
SABETI:这是我们所说的“从变异到功能”的界面,来自数学、计算机科学和计算生物学的个人与病毒学家、分子生物学家、传染病研究人员和临床医生聚集在一起。从长凳到床边,你看到了模式,并对它们产生了兴趣。
宪报:很明显,预测变异以及哪些变异将占主导地位的能力是很重要的。你对这款机型的前景有什么看法?
SABETI:这个领域的圣杯通常是能够从一开始就预测哪些突变是重要的,它们的影响是什么,本质上是微生物将如何适应。要做到这一点,我们需要这些庞大的模型来真正研究病毒和微生物基因组,当你看到不同的突变足够多的时候,开始找出模式和潜在的逻辑。我认为我们可以开始了解适应是如何发生的,以及我们应该如何在制定对策时解决它,但这将需要大量的数据。每当有人问:“我们是否产生了太多的数据?”我的回答是:绝对没有。我们真的应该做到这一点,对感染中检测到的每一个微生物基因组进行测序,因为有些事情我们甚至不知道是可能的,因为我们没有数据。
进一步探索
这个故事是由哈佛大学报》哈佛大学的官方报纸。欲了解更多大学新闻,请访问Harvard.edu.