研究人员利用全基因组测序扩展和升级1000个基因组计划资源
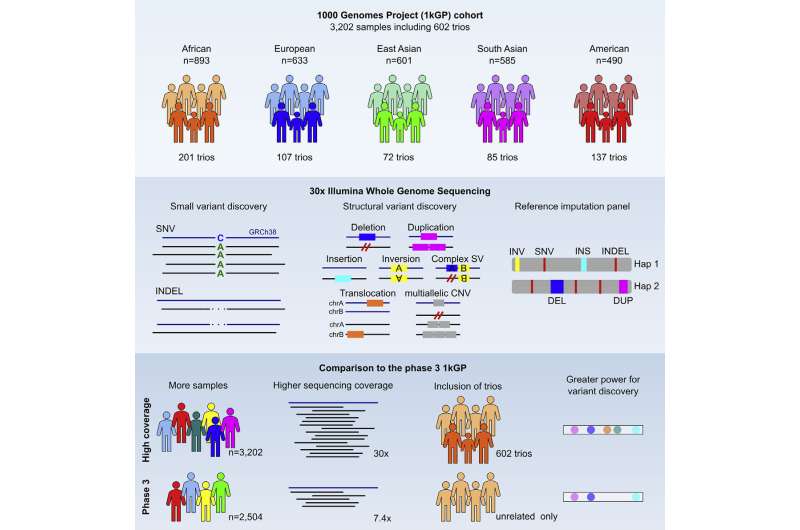
七年前,1000基因组计划(1kGP)发布了一个开放获取的资源,主要基于低覆盖率的全基因组测序(WGS)数据,这些数据来自代表世界五大洲的26个种群的2504个个体,这是WGS首次大规模提供人类遗传变异目录。
现在,纽约基因组中心(NYGC)的研究人员与马萨诸塞州总医院、耶鲁大学和人类基因组结构变异联盟(HGSVC)的研究小组合作,将1kGP资源扩展到几乎所有的亲子三人组,以及原始样本,并使用Illumina NovaSeq仪器对它们进行高覆盖率测序。这项研究发表在细胞,对扩大的1kGP队列的高覆盖率WGS数据进行了全面分析,该队列目前由3202个样本组成,其中包括602个三组。
“1000个基因组计划队列是如此宝贵的资源,我们认为它将对社区有用,使测序与最新版本的短读技术同步,同时增加以前被忽略的家族样本的丰富性,”纽约gc计算生物学科学主任、该研究的高级作者迈克尔·佐迪博士解释说。
利用最先进的方法和算法,NYGC的研究人员对来自淋巴母细胞系(LCLs;即,来自外周血的永生化人类B细胞)从扩展队列到30倍基因组覆盖的目标深度。接下来,研究小组进行了单核苷酸测序变体(SNV)和短插入和删除(INDEL)调用,其中包括从序列数据相对于人类基因组对队列中所有样本中发现的变异位点进行参考和基因分型。
此外,哈佛医学院、布罗德研究所和马萨诸塞州总医院Michael Talkowski博士团队与耶鲁大学Ira Hall博士团队和华盛顿大学医学院以及HGSVC合作,通过整合多种分析方法,在3202个1kGP样本中发现了一组全面的结构变异(SVs),并对其进行了基因分型。
总体而言,该研究表明,在变异调用的发现能力和精度方面都有了显著提高,特别是在罕见的snv以及跨越频谱的indel和sv中,这些都是以前低覆盖率测序无法获得的。
原始1kGP资源的一个重要方面是将其用作变体imputation的参考组,即,基于从参考组中学习的群体中通常共同遗传的变体分组,在稀疏的、基于阵列的样本中对未观察到的基因型进行统计推断,这促进了大量全基因组关联研究(GWAS)。现在,随着原始资源的扩展,该团队升级了参考imputation面板,以包括通过高覆盖率WGS和trio family发现的更多变体。
NYGC的高级生物信息学科学家Marta Byrska-Bishop博士解释说:“新的imputation小组包括更多的位点,特别是许多更常见的INDELs和SVs,从而扩大了GWAS可获得的变异的数量,考虑到非snv变异的大效应量,很可能能够发现新的遗传关联,有助于确定致病变异。”
所有原始序列数据和变体调用集在测序完成后立即通过几个基因组数据存储库向公众发布,包括由欧洲分子生物学实验室(EMBL-EBI)欧洲生物信息学研究所的共同作者维护的国际基因组样本资源(IGSR)。
“我们的目标是让这一公共资源成为未来群体遗传研究和方法发展的基准,”麻省总医院基因组医学中心博士后赵雪芳博士补充道,他是该研究的共同第一作者。
这些数据已经引起了遗传学和基因组学界的兴趣。由于1kGP样品的完全开放获取性质,这种情况可能会持续数年,与大多数新出现的WGS工作不同,它是同意公开分发的遗传不受访问或使用限制的数据。
进一步探索