研究人员开发了一种改进癌症治疗的算法
UVA健康癌症中心的研究人员开发了一种算法,可以通过快速、轻松地识别将从名为激酶抑制剂的强效癌症药物中受益的患者来改善癌症护理。该算法对患者还有其他的诊断益处。
激酶抑制剂是最常见的癌症联邦食品和药物管理局批准的药物它们对合适的病人非常有效,但并不是对所有人都有效。UVA的算法提供了一种新的、更好的方法来确定谁将受益——这是为个人量身定制的精准医疗向前迈出的重要一步。
“我们对这个算法感到非常兴奋,它比现有的要求和假设更少的方法表现得更好,使它更适用于从肿瘤的单个快照了解癌症状态,”弗吉尼亚大学生物医学工程系的研究员Kristen M. Naegle说,该学系是弗吉尼亚大学医学院和工程学院的一个联合项目。“将这种方法与现有的生物标志物相结合癌症诊断可能会帮助我们更好地调整疗法,设计新的联合疗法,预测治疗反应,设计更好的临床试验。”
为更好的癌症治疗KSTAR
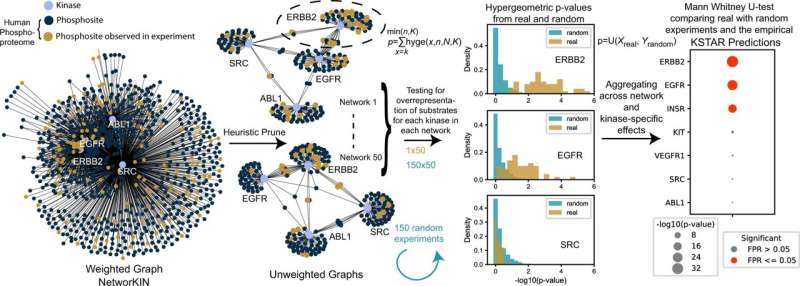
奈格尔和他的同事着手克服现有方法的局限性,以确定哪些患者可能受益于激酶抑制剂.大多数这些方法都需要难以获得、有时不可靠的量化细胞内“磷酸化位点”的信息。然而,UVA的新方法不需要所有这些复杂的测量。相反,它可以根据其他可用数据预测关键信息。
这使得算法可以为被称为激酶的单个酶产生一个特定的“KSTAR评分”。医生可以使用这些分数来确定哪些患者对激酶抑制剂有反应,帮助指导最佳的治疗选择。激酶抑制剂广泛应用于某些类型的血癌、乳腺癌和肺癌等。这些药物通常与化疗、放疗或手术等其他治疗方法联合使用。
在测试他们的新算法时,奈格尔和她的合作者发现它在不同的组织类型上都能可靠地工作,这表明它对许多类型的癌症都有用。
作为一个额外的好处,KSTAR可以作为诊断工具弗吉尼亚大学的研究人员报告说。在他们的测试中,KSTAR能够确定一个患有乳腺癌并不像医生之前认为的那样是HER2阳性。her2阳性乳腺癌可受益于her2靶向治疗激酶但her2阴性肿瘤不会,her2抑制会带来额外的并发症。
此外,研究人员发现,在目前的临床方法中,约20%的HER2阴性患者实际上具有HER2特征,这表明他们可以从HER2靶向治疗中受益——这是一种目前没有提供给他们的治疗方法。在医生和病人讨论治疗方案时,这类信息可能是无价的。
纳格尔说:“我们正在与各种癌症的研究人员团队合作,以确定KSTAR何时以及如何帮助确定患者对治疗的反应。”“我们希望能够帮助更好地确定在正确的时间对正确的病人进行正确的治疗,以获得更好的结果病人."
内格尔和她的合作者已经将他们的新算法免费提供。他们的研究结果发表在自然通讯.