人工智能可以加速新药的发现

谢菲尔德大学(University of Sheffield)和阿斯利康(AstraZeneca)的研究人员合作开发了一种可以降低成本、加速新药发现的人工智能。
这项新技术是由谢菲尔德大学计算机科学系的陆海平教授和他的博士生白培珍,以及阿斯利康大学的菲利普·米耶科维奇博士和比诺·约翰博士共同开发的自然机器智能.
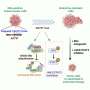
这项研究表明,名为DrugBAN的人工智能可以预测候选人是否合适药物会与体内的靶蛋白分子相互作用人体.
根据2023年2月2日发表的论文,可以预测药物是否会达到预期目标的人工智能已经存在,但谢菲尔德大学和阿斯利康大学的研究人员开发的技术可以更准确地做到这一点,并提供有用的见解,帮助科学家了解药物如何在分子水平上与蛋白质伙伴发生作用。
人工智能有可能告知一种药物是否会成功地与预期的癌症相关蛋白质或者一种候选药物是否会与体内的非预期靶点结合,从而给患者带来不良的副作用。
人工智能被训练来学习人体蛋白质的亚结构以及药物化合物的亚结构。然后,该技术了解这些子结构如何相互作用,并据此预测新药可能的表现。
谢菲尔德大学机器学习教授陆海平表示:“我们设计人工智能有两个主要目标。首先,我们希望人工智能能够在更细的尺度上捕捉药物与靶标的相互作用,因为这可以提供有用的生物学见解,帮助研究人员在分子水平上理解这些相互作用。其次,我们希望该工具能够预测这些与新药或靶标的相互作用,以帮助加快整体预测过程。我们今天发表的研究表明,我们的人工智能模型可以做到这两点。”
人工智能设计的关键是模型如何学习成对的子结构相互作用——药物化合物的子结构和体内蛋白质之间可能发生的多种相互作用。然而,市场上大多数现有的药物预测AI都是从药物和蛋白质的整体表示中学习的,这些表示不能捕获它们的亚结构,因此提供的见解不太有用。
在AI开发的下一阶段,该团队计划使用更深入的化合物和蛋白质结构数据,使AI更加准确。
阿斯利康临床药理学和安全科学(CPSS)数据科学总监Bino John博士说:“DrugBAN的一个关键新颖之处在于它依赖于双线性注意力网络,使其能够同时从药物和靶标的子结构中学习相互作用。我们还做了源代码免费向公众开放,希望这将支持更多的人工智能方法,继续加速药物发现。”
使用传统方法进行药物发现和开发可能会非常困难,因为开发时间很长,需要花费大量资金。然而,药物发现进程有可能大大加快;随着人工智能的进步数字技术在美国,研究人员正在寻找新的方法来确定药物可能与我们体内的哪些蛋白质相互作用。
尼克·布朗,成像和数据分析阿斯利康(AstraZeneca)的CPSS表示:“看到这篇论文我真的很兴奋,特别是因为与其他方法不同,DrugBAN同时使用双线性注意力网络从候选药物和它们的靶标中学习,并且明确地设计为概括问题。”
谢菲尔德大学计算机科学系主任盖伊·布朗教授补充说:“我们在谢菲尔德的研究强烈地出于对人们生活产生积极影响的愿望,我们认为与阿斯利康等行业领导者的互动对实现这一使命至关重要。
“这是一项令人兴奋的研究,有望在治疗设计方面取得重大进展。该方法的独特之处在于它注重可解释性,使人类专家能够从人工智能系统产生的见解中受益。”
更多信息:Peizhen Bai等,具有域自适应的可解释双线性注意网络改进药物-目标预测,自然机器智能(2023)。DOI: 10.1038 / s42256 - 022 - 00605 - 1